DBSCAN的全部英文是Density-Based Spatial Clustering of Applications with Noise,中文是“基于密度的带有噪声的空间聚类”。DBSCAN是一个比较有代表性的基于密度的聚类算法,与划分和层次聚类方法不同,它将簇定义为密度相连的点的最大集合,能够把具有足够高密度的区域划分为簇,并可在噪声的空间数据库中发现任意形状的聚类。
该算法利用基于密度的聚类的概念,即要求聚类空间中的一定区域内(用Eps定义出的半径)所包含对象(点或其他空间对象)的数目不小于某一给定阈值(用MinPts定义的聚类点数)。
DBSCAN算法的显著优点是聚类能够 有效处理噪声点 和发现 任意形状的空间聚类。但是由于它直接对整个数据库进行操作且进行聚类时使用了一个全局性的表征密度的参数,因 此也具有两个比较明显的弱点:
- 对于高维问题, 基于Eps和MinPts密度定义是个很大问题
- 当簇的密度变化太大时,聚类结果较差
- 当数据量增大时,要求较大的内存支持,I/0消耗也很大
DBSCAN算法的目是基于密度寻找被低密度区域分离的高密度,以此来实现不同样本点的聚类。跟传统聚类算法如K-Means相比,它具有以下优点:
- 原始数据集的分布没有明显要求,因此数据集适用性更广,尤其是非凸装、圆环形等异形簇分布的识别较好;
- 与K-Means相比,无需指定聚类数量,对结果的先验要求不高;
- 由于DBSCAN可区分核心对象、边界点和噪音点,因此对噪声的过滤效果好。
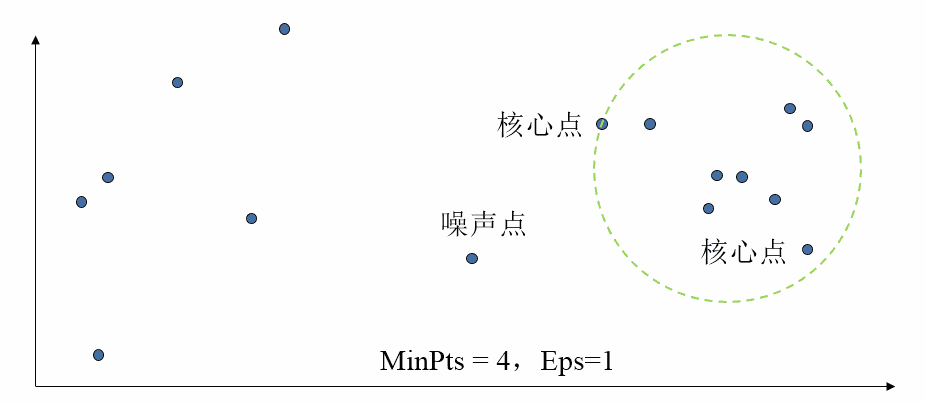
基于密度的定义,可将所有样本点分为三类:
- 稠密区域内部的点(核心点):在半径Eps内含有超过MinPts数目的点。
- 稠密区域边缘上的点(边界点):在半径Eps内点的数量小于MinPts(即不是核心点),但是其邻域内包含至少一个核心点。
- 稀疏区域中的点(噪声或背景点): 任何不是核心点或边界点的点。
DBSCAN应用
以下使用Python机器学习库SKlearn中的DBSCAN来进行演示演示,目标是对随机生成的数据集进行聚类。 `
#coding:utf-8
import numpy as np
from sklearn.cluster import DBSCAN
from sklearn import metrics
from sklearn.datasets.samples_generator import make_blobs
from sklearn.preprocessing import StandardScaler
import matplotlib.pyplot as plt
# 生成训练数据
centers = [[1, 1], [-1, -1], [1, -1]]
X, labels_true = make_blobs(n_samples=750, centers=centers, cluster_std=0.4, random_state=0)
X = StandardScaler().fit_transform(X)
# 训练DBSCAN
db = DBSCAN(eps=0.3, min_samples=10).fit(X)
core_samples_mask = np.zeros_like(db.labels_, dtype=bool)
core_samples_mask[db.core_sample_indices_] = True
labels = db.labels_
new_X = np.column_stack((X, labels))
# 如果存在噪音则去除
n_clusters_ = len(set(labels)) - (1 if -1 in labels else 0)
print('Estimated number of clusters: %d' % n_clusters_)
print("Homogeneity: %0.3f" % metrics.homogeneity_score(labels_true, labels))
print("Completeness: %0.3f" % metrics.completeness_score(labels_true, labels))
print("V-measure: %0.3f" % metrics.v_measure_score(labels_true, labels))
print("Adjusted Rand Index: %0.3f"
% metrics.adjusted_rand_score(labels_true, labels))
print("Adjusted Mutual Information: %0.3f"
% metrics.adjusted_mutual_info_score(labels_true, labels))
print("Silhouette Coefficient: %0.3f"
% metrics.silhouette_score(X, labels))
print("Top 10 samples:",new_X[:10])
# 图形展示
unique_labels = set(labels)
colors = plt.cm.Spectral(np.linspace(0, 1, len(unique_labels)))
for k, col in zip(unique_labels, colors):
if k == -1:
# 噪音显示为黑色.
col = 'k'
class_member_mask = (labels == k)
xy = X[class_member_mask & core_samples_mask]
plt.plot(xy[:, 0], xy[:, 1], 'o', markerfacecolor=col,
markeredgecolor='k', markersize=14)
xy = X[class_member_mask & ~core_samples_mask]
plt.plot(xy[:, 0], xy[:, 1], 'o', markerfacecolor=col,
markeredgecolor='k', markersize=6)
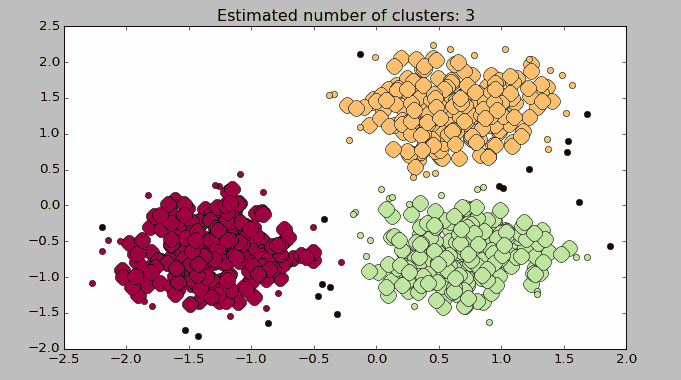
plt.title('Estimated number of clusters: %d' % n_clusters_)
plt.show()
运行结果如下: `
Estimated number of clusters: 3
Homogeneity: 0.953
Completeness: 0.883
V-measure: 0.917
Adjusted Rand Index: 0.952
Adjusted Mutual Information: 0.883
Silhouette Coefficient: 0.626
('Top 10 samples:', array(
[[ 0.49426097, 1.45106697, 1. ],
. [-1.42808099, -0.83706377, 0. ],
. [ 0.33855918, 1.03875871, 1. ],
. [ 0.11900101, -1.05397553, 2. ],
. [ 1.1224246 , 1.77493654, 1. ],
. [-1.2615699 , 0.27188135, 0. ],
. [-1.30154775, -0.76206203, 0. ],
. [ 0.58569865, -0.33910463, 2. ],
. [ 1.08247212, 0.8868554 , 1. ],
. [ 1.01416668, 1.34114022, 1. ]]))
DBSCAN可配置的参数如下,重点是半径eps和密度min_samples的定义: `
class sklearn.cluster.DBSCAN(eps=0.5, min_samples=5, metric='euclidean', algorithm='auto', leaf_size=30, p=None, random_state=None)
DBSCAN应用场景:
- 数据集聚类,如会员聚类;
- 图像切割;
尾巴
DBSCAN的最大优势是它对于原始数据集的分布几乎没有要求,并且对噪音不敏感,因此,它的模型适应性和鲁棒性相对较强;
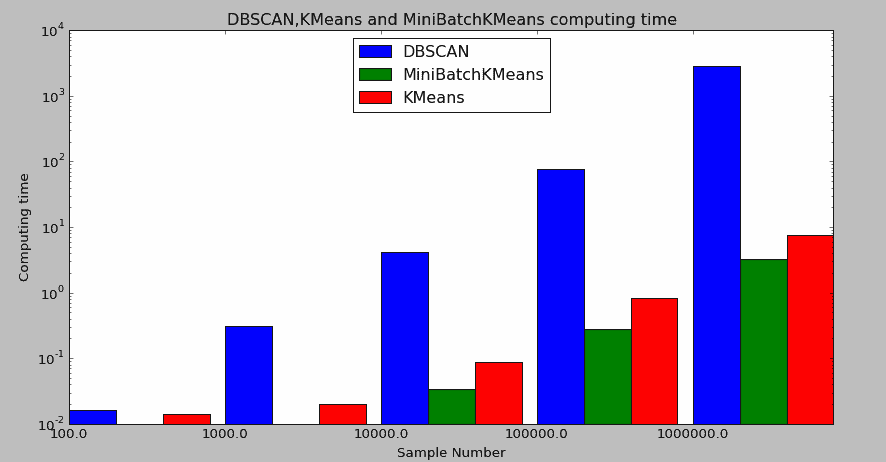
但是,它对于大数据集的处理速度只能算是一般,以下是分别针对100,1000,10000,100000,1000000样本量进行聚类(类别数为三,二维),得到三类算法KMeans、MiniBatchKMeans和DBSCAN的聚类用时。
由于三者计算用时差异比较大,因此取对数进行图形对比。
从结果可以看出,MiniBatchKMeans由于是KMeans的优化版本,因此用时少是毫无疑问的,重点是KMeans和DBSCAN的对比发现,DBSCAN在对100万数据聚类时用时竟然达到40多分钟。
 `
`
('DBSCAN Time:', [[100, 0.015000104904174805], [1000, 0.2969999313354492], [10000, 3.9000000953674316], [100000, 69.59299993515015], [1000000,2854.7410000000]])
('MiniBatchKMeans Time:', [[100, 0.016000032424926758], [1000, 0.016000032424926758], [10000, 0.031000137329101562], [100000, 0.20300006866455078], [1000000, 1.748000144958496]])
('KMeans Time:', [[100, 0.014999866485595703], [1000, 0.015000104904174805], [10000, 0.07799983024597168], [100000, 0.7639999389648438], [1000000, 6.769999980926514]])
`